10.4 Manipulação de dados
Com os dados arrumados, a próxima etapa é a manipulação dos dados. O pacote dplyr oferece um conjunto de funções que facilita as operações mais comuns para lidar com dados retangulares de uma forma bem pensada.
Os verbos fundamentais desta gramática de manipulação de dados são:
select(), para selecionar variáveis;filter(), para filtrar observações;arrange(), para classificar variáveis;mutate(), para criar e transformar variáveis;group_by(), para agrupar observações;summarise(), para resumir os dados com medidas estatísticas descritivas;
Estes verbos possuem uma sintaxe consistente com uma sentença gramatical:
verbo(sujeito, complemento)
função(dados, z = x + y)
- o
verboé a função do dplyr; - o
sujeito(dados) é quem sofre a ação e é sempre o primeiro argumento, nomeado (.data); - o
complementosão expressões que podem ser usadas como argumentos (o que é representado pela reticência...no segundo argumento); isso ficará mais claro nos exemplos mais a frente;
Os verbos listados anteriormente possuem versões equivalentes na base do r rblue. Então, por que usar o dplyr ?
-
é muito mais rápido de se aprender, com poucas funções (ou verbos) nomeadas intuitivamente;
-
as funções do dplyr são mais rápidas (parte dos códigos são programados em C++);
-
trabalha bem com dados arrumados e também com sistemas de banco de dados
-
as funções foram projetadas para trabalharem juntas na solução diversos problemas de processamento de dados;
10.4.1 Códigos como fluxogramas
A manipulação de dados requer uma organização apropriada do código. A medida que novas etapas do fluxo de trabalho vão sendo implementadas o código expande-se. As etapas vão sendo implementadas de forma sequencial, combinando funções que geram saídas que servirão de entrada para outras funções na cadeia de processamento.
Essa é justamente a ideia do operador pipe %>%: passar a saída de uma função para outra função como a entrada dessa função por meio de uma seqüência de etapas. O operador %>% está disponível no ![]() através do pacote magrittr.
através do pacote magrittr.
Os pacotes tidyverse integram-se muito bem com o %>%, por isso ele é automaticamente carregado com o tidyverse. Vamos ilustrar as vantagens de uso do %>% com exemplos a seguir.
10.4.1.1 Vantagens do %>%
O exemplo a baixo mostra uma aplicação simples do %>% para extrair a raiz quadrada de um número com a função base::sqrt()e a extração do segundo elemento de um vetor com a função dplyr::nth() (uma função alternativa aos colchetes []).
# chamada tradicional de uma função
sqrt(4)
nth(5:1, 2)
# chamada de uma função com %>%
4 %>% sqrt()
5:1 %>% nth(2)Ambas formas realizam a mesma tarefa e com mesmo resultado e o benefício do %>% não fica evidente. Entretanto, quando precisamos aplicar várias funções as vantagens ficam mais óbvias.
No código abaixo tente decifrar o objetivo das operações no vetor x.
x <- c(1, 3, -1, 1, 4, 2, 2, -3)
x
#> [1] 1 3 -1 1 4 2 2 -3
nth(sort(cos(unique(x)), decreasing = TRUE), n = 2)
#> [1] 0.5403023Talvez com o código identado fique mais claro:
nth( # 4
sort( # 3
cos( # 2
unique(x) # 1
),
decreasing = TRUE
),n = 2
)O código acima está aninhando funções e isso leva a uma dificuldade de ler por causa da desordem. Para interpretá-lo precisamos fazer a leitura de dentro para fora:
- mantém somente os valores únicos de x
- calcula o cosseno do resultado de (1)
- coloca em ordem decrescente o resultado de (2)
- extrai o 2° elemento do resultado de (3)
Conclusão: o objetivo era obter o segundo maior número resultante do cosseno do vetor numérico x.
A versão usando pipe é:
x %>%
unique() %>% # 1
cos() %>% # 2
sort(decreasing = TRUE) %>% # 3
nth(n = 2) # 4Dessa forma, o código fica mais simples, legível e explícito. Por isso, daqui para frente, nós utilizaremos extensivamente o operador %>% para ilustrar os verbos do dplyr e suas combinações.
No exemplo anterior nós introduzimos a função dplyr::nth(). Ela é equivalente ao operador colchetes [ da base do R. Se a <- 5:1 então as instruções abaixo produzem resultados equivalentes:
a[2]; nth(a, 2)
#> [1] 4 #> [1] 4
10.4.1.2 O operador . como argumento
Uma representação mais explícita do código usado na cadeia de funções acima, seria com a inclusão do operador . e os nomes dos argumentos das funções:
x %>%
unique(x = .) %>% # 1
sort(x = ., decreasing = TRUE) %>% # 2
cos(x = .) %>% # 3
nth(x = ., n = 2) # 4O tempo a mais digitando é compensado posteriormente quando o você mesmo futuramente tiver que reler o código. Essa forma enfatiza com o . que o resultado à esquerda é usado como entrada para função à direita do %>%.
Mas nem todas funções do ![]() foram construídas com os dados de entrada no primeiro argumento. Essa é a deixa para outra funcionalidade do
foram construídas com os dados de entrada no primeiro argumento. Essa é a deixa para outra funcionalidade do . que é redirecionar os dados de entrada para a posição adequada naquelas funções. Uma função que se encaixa neste caso é a base::grep() que detecta uma expressão regular num conjunto de caracteres (strings).
adverbs <- c("ontem", "hoje", "amanhã")
grep(
pattern = "h",
x = adverbs,
value = TRUE
)O código acima seve para retornar os elementos do vetor dias que contenham a letra h. No entanto os dados de entrada da base::grep() são esperados no 2° argumento (x). Para redirecioná-los para essa posição dentro de uma cadeia de funções com %>%, colocamos o operador . no 2° argumento da função:
adverbs %>%
grep(
pattern = "h",
x = .,
value = TRUE
)10.4.2 Seleção de variáveis
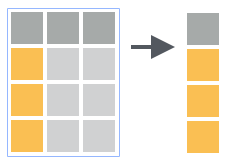
Para selecionar somente variáveis de interesse em uma tabela de dados podemos usar a função dplyr::select(.data, ...). Nos dados clima_rs_tbl se desejamos selecionar apenas as colunas estacao e tmax aplicamos a dplyr::select() com o 2° argumento listando as colunas que desejamos selecionar:
select(clima_rs_tbl, estacao, tmax)O resultado é um subconjunto dos dados originais contendo apenas as colunas nomeadas nos argumentos seguintes aos dados de entrada.
A função dplyr::select() possui funções auxiliares para seleção de variáveis:
clima_rs_tbl %>%
# as variáveis entre uf e tmax
select(., uf:tmax) %>%
head(., n = 3)
clima_rs_tbl %>%
# todas variáveis menos as entre codigo:uf
select(., -(codigo:uf)) %>%
head(., n = 3)
clima_rs_tbl %>%
# ordem inversa das variáveis
select(., tmax:codigo) %>%
head(., n = 3)
clima_rs_tbl %>%
# nomes que contenham a letra "a"
select(., contains("a")) %>%
head(n = 3)
clima_rs_tbl %>%
# variáveis que iniciam com "c"
select(., starts_with("c")) %>%
head(., n = 3)
clima_rs_tbl %>%
# usando um vetor de caracteres
select(., any_of(c("estacao", "uf"))) %>%
head(., n = 3)
clima_rs_tbl %>%
# combinações
select(., -uf, ends_with("o")) %>%
head(., n = 3)
clima_rs_tbl %>%
# variáveis que inciam com letras minúsculas e com 4 caracteres
select(., matches("^[a-z]{4}$")) %>%
head(., n = 3)
# variáveis numéricas
clima_rs_tbl %>%
select(is.numeric)O último exemplo usa uma expressão regular (regex). Regex é uma linguagem para descrever e manipular caracteres de texto. Há livros sobre este assunto e diversos tutorias sobre regex no R. Para saber mais sobre isso veja o capítulo sobre strings do livro de H. Wickham and Grolemund (2017). Conhecendo o básico, você poupará tempo automatizando a formatação de caracteres de texto.
Veja mais funções úteis para seleção de variáveis em ?dplyr::select.
10.4.3 Seleção de observações
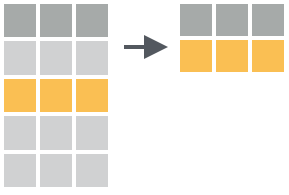
A filtragem de observações geralmente envolve uma expressão que retorna valores lógicos ou as posições das linhas selecionadas (como a função which()).
A função dplyr::filter() permite filtrar observações de um data frame correspondentes a alguns critérios lógicos. Estes critérios podem ser passados um de cada vez ou com um operador lógico (e: &, ou: |). Veja abaixo alguns exemplos de filtragem de observações:
- linhas correspondentes ao
codigoda estação 83936.
clima_rs_tbl %>%
filter(codigo == 83936)- linhas da variável
estacaoque contenham o vetor caracterelitoraneas.
litoraneas <- c("Torres",
"Guaporé")
clima_rs_tbl %>%
filter(estacao %in% litoraneas)- observações com
tmaxacima de 10% da média
filter(clima_rs_tbl, tmax > 1.1*mean(tmax))- observações com
tmaxeprecacima de suas médias
clima_rs_tbl %>%
filter(
tmax > mean(tmax),
prec > mean(prec)
)
# equivalente a
#clima_rs %>%
# filter(tmax > mean(tmax) & prec > mean(prec))- observações cuja variável
estacaotem a palavra "Sul"
# estações com "Sul" no nome
clima_rs_tbl %>%
filter(str_detect(estacao, "Sul"))
O exemplo acima é mais uma operação com caracteres onde foi usada a função stringr::str_detect() para detectar os elementos da variável do tipo caractere que contenham o termo "Sul". O pacote stringr (Hadley Wickham 2018) fornece funções para casar padrões de caracteres de texto e os nomes das funções são fáceis de lembrar. Todos começam com str_ (de string) seguido do verbo, p.ex.:
str_replace_all(
string = c("abc", "lca"),
pattern = "a",
replacement = "A"
)
#> [1] "Abc" "lcA"
A seleção de observações também pode ser baseada em índices passados para função dplyr::slice() que retorna o subconjunto de observações correspondentes. Abaixo vejamos alguns exemplos de filtragem de linhas baseada em índices ou posições:
# linhas 2 e 4
clima_rs_tbl %>%
slice(., c(2, 4))
# última linha
clima_rs_tbl %>%
slice(., n())
# exlui da última à 3a linha
clima_rs_tbl %>%
slice(., -(n():3))
# linhas com tmax > 26
clima_rs_tbl %>%
slice(., which(tmax > 26))
# linhas com tmax mais próxima a média de tmax
clima_rs_tbl %>%
slice(., which.min(abs(tmax - mean(tmax))))10.4.4 Reordenando dados
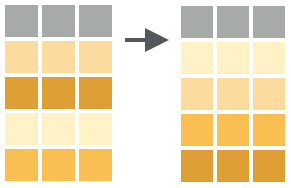
As vezes é útil reordenar os dados segundo a ordem (crescente ou decrescente) dos valores de uma variável. Por exemplo, os dados clima_rs_tbl podem ser arranjados em ordem decrescente da precipitação anual, conforme abaixo.
clima_rs_tbl %>%
arrange(., desc(prec)) %>%
head(., n = 3)A função dplyr::arrange() por padrão ordena os dados em ordem crescente. A função dplyr::desc() ordena os valores da variável em ordem descendente.
Os dados ordenados pela tmax, ficam da seguinte forma:
clima_rs_tbl %>%
arrange(., tmax) %>%
head(., n = 3)10.4.5 Criando e renomeando variáveis

Uma nova variável pode ser adicionada aos dados através da função dplyr::mutate(). A tmax expressa em Kelvin pode ser adicionada aos dados clima_rs_tbl, com:
clima_rs_tbl %>%
# tmax em Kelvin
mutate(., tmaxK = tmax + 273.15) %>%
# só as colunas de interesse
select(., contains("tmax")) %>%
# 3 primeiras linhas
head(., n = 3)Podemos renomear variáveis com a função dplyr::rename().
clima_rs_tbl %>%
rename(.,
"id" = codigo,
"site" = estacao,
"temp_max" = tmax,
"precip" = prec
) %>%
head(., n = 3)Podemos sobrescrever variáveis e recodificar seus valores, conforme o exemplo abaixo. A variável site será corrigida, de forma os valores iguais a "A803" sejam substituídos por "A003".
prec_anual_corr <- prec_anual %>%
mutate(
site = recode(site, A803 = "A003")
)
tail(prec_anual_corr, n = 4)Podemos preencher os valores faltantes de uma variável por um valor prescrito, por exemplo baseado na média de outras observações, ou nos valores prévios, ou posteriores. Variáveis podem ser derivadas das variáveis sendo criadas dentro da dplyr::mutate().
# preenchendo prec faltante pela média
prec_anual_comp %>%
mutate(.,
prec = replace_na(prec,
mean(prec, na.rm = TRUE)
),
ndias = ifelse(ano %% 4 == 0,
366,
365),
# intensidade de ndias, criada na linha acima
intensidade = prec / ndias
)
prec_anual_comp %>%
# preenche com a observação prévia
fill(prec, .direction = "down")
prec_anual_comp %>%
# preenche com a observação posterior
fill(prec, .direction = "up")10.4.6 Agregando observações
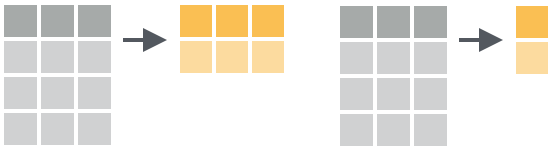
A função dplyr::summarise() (ou dplyr::sumarize()) agrega valores de uma variável e os fornece para uma função que retorna um único resultado. O resultado será armazenado em um data frame.
Por exemplo, qual a prec média anual do RS?
clima_rs_tbl %>%
summarise(
.,
prec_med = mean(prec)
)Se você só quer o valor (ou o vetor), ao invés de um data frame, pode usar a função dplyr::pull():
clima_rs_tbl %>%
summarise(
.,
prec_med = mean(prec)
) %>%
pull()Podemos aplicar uma ou mais funções a mais de uma variável usando dplyr::summarise_at():
clima_rs_tbl %>%
summarise_at(
.,
.vars = vars(prec, tmax),
.funs = list(mn = min, md = median, mx = max),
na.rm = TRUE
)Observações repetidas devem ser removidas dos dados antes de qualquer cálculo. Suponha os dados abaixo:
prec_anual_comp_rep <-
prec_anual_comp %>%
mutate(
site = recode(site, A803 = "A003"),
ano = NULL
) %>%
# preenche com a observação posterior
fill(., prec, .direction = "up")
prec_anual_comp_repPara desconsiderar linhas duplicadas nos dados usamos a função dplyr::distinct():
# remove observações repetidas
prec_anual_comp_rep %>%
distinct(site, prec)A função dplyr::count() é útil para obter a frequência de ocorrência de uma variável ou da combinação de variáveis.
prec_anual_comp_rep %>%
count(site)
prec_anual_comp_rep %>%
count(site, prec)10.4.7 Agrupando observações
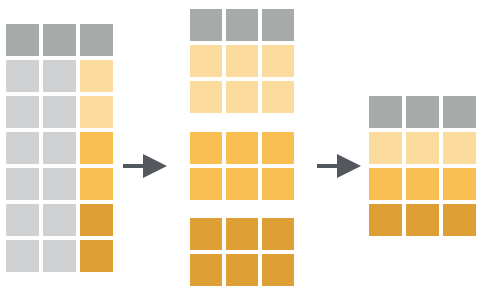
Frequentemente temos que agrupar observações em categorias ou grupos para realizar uma análise estatística. A função dplyr::group_by() é uma função silenciosa que separa (invisivelmente) as observações em categorias ou grupos. A única mudança ao aplicar a dplyr::group_by() à um data frame é a indicação da variável agrupada e o seu número de grupos na saída do console. No exemplo a seguir vamos agrupar os dados prec_anual_tbl por site e teremos 4 grupos para esta variável.
prec_anual_tbl %>%
group_by(site)A grande funcionalidade da dplyr::group_by() surge quando combinada com a função dplyr::summarise(), o que nos permite obter resumos estatísticos para cada grupo da variável.
Por exemplo a chuva anual média por site (estação meteorológica) é obtida com o seguinte código:
prec_anual_tbl %>%
group_by(., site) %>%
summarise(., prec_med = mean(prec))A prec média para cada ano e o número de anos utilizados em seu cálculo é obtida por:
prec_anual_tbl %>%
group_by(., ano) %>%
summarise(
.,
prec_med = mean(prec),
nobs = n()
)A função n() conta quantas observações temos em um subconjunto dos dados.
Os grupos podem ser compostos de mais de uma variável. Para o exemplo com os dados prec_anual_long;
prec_anual_longpodemos obter a média por variavel e site, fazendo:
por_site_var <- prec_anual_long %>%
group_by(site, variavel) %>%
summarise(
media = mean(medida, na.rm = TRUE)
) %>%
arrange(variavel, site)
por_site_varCom o conjunto de verbos exemplificados você agora é capaz de realizar as tarefas mais comuns de manipulação de dados tabulares de forma clara e confiável.
Há mais funções úteis disponíveis no pacote dplyr e você é encorajado a descubrí-las.
10.4.8 Combinação de dados
O processamento de dados frequentemente envolve a manipulação de diversas tabelas de dados. Ás vezes precisamos juntar dados de diferentes fontes, formar uma tabela única de dados com o período em comum à elas, ou combiná-las para compará-las.
A combinação de 2 data frames, com observações similares, que tem variáveis diferentes e algumas em comum é uma tarefa muito comum na manipulação de conjuntos dados. Este tipo de operação é chamada de junção (do termo em inglês join) de tabelas . O pacote dplyr possui uma gama de funções do tipo join para combinar data frames, genericamente representadas por <tipo>_join(), onde <tipo> pode ser substituído por dplyr::full_join(), dplyr::inner_join(), dplyr::left_join(), dplyr::right_join().
Essas funções combinam informação em dois data frames baseada na unificação de valores entre as variáveis que ambos compartilham.
Vamos considerar os dados clima_rs_tbl e metadados_rs. Para melhor compreensão do exemplo vamos remover algumas variáveis.
# normais climatológicas das estaçõess
clima_rs_tbl <- clima_rs_tbl %>%
select(-(estacao:uf))
head(clima_rs_tbl)
# metadados das estações convertidos para tibble
metadados_rs <- metadados_rs %>%
as_tibble()
head(metadados_rs)A variável comum às duas tabelas é:
var_comum <- names(clima_rs_tbl) %in% names(metadados_rs)
names(clima_rs_tbl)[var_comum]Vamos comparar os valores da variável codigo em cada tabela de dados para verificar se todos valores contidos em uma tabela também estão presentes na outra e vice-versa.
Para saber se algum valor da variável codigo da tabela clima_rs_tbl não está contido na tabela metadados_rs podemos usar o seguinte código:
# algum codigo não está presente na tabela metadados_rs
clima_rs_tbl %>%
filter(., ! codigo %in% metadados_rs$codigo ) %>%
select(., codigo)Não há nenhum caso.
Analogamente, vamos verificar se algum valor da variável codigo dos metadados_rs não está contido em clima_rs_tbl.
# algum codigo não está presente na tabela metadados_rs
metadados_rs %>%
filter(., ! codigo %in% clima_rs_tbl$codigo )%>%
select(., codigo)Obtemos que 7 valores da variável codigo dos metadados_rs que não estão presentes na tabela clima_rs_tbl. Portanto, não há valores de tmax e prec para essas observações.
Suponha agora que desejássemos visualizar a variação espacial da precipitação (prec) ou da temperatura máxima do ar (tmax) climatológica. Precisaríamos além dessas variáveis, as coordenadas geográficas das estações meteorológicas para plotar sua localização espacial. As coordenadas lon e lat da metadados_rs podem ser combinadas com clima_rs_tbl em uma nova tabela (clima_rs_comb), usando a função dplyr::full_join():
clima_rs_comb <- full_join(
x = clima_rs_tbl,
y = metadados_rs,
by = "codigo")
clima_rs_combDa inspeção das últimas linhas de clima_rs_comb verificamos que o resultado é uma tabela que contém todos valores da variável codigo das duas tabelas. Os valores das variáveis prec e tmax, para as observações da variável codigo sem valores ( na metadados_rs) são preenchidos com NA.
Se a combinação de interesse for nas observações em comum entre as tabelas, usaríamos:
clima_rs_intersec <- inner_join(
x = metadados_rs,
y = clima_rs_tbl,
by = "codigo"
)
clima_rs_intersecPara obter uma tabela com as observações diferentes entre as duas tabelas, usamos:
clima_rs_disj <- anti_join(
x = metadados_rs,
y = clima_rs_tbl,
by = "codigo"
)
clima_rs_disjO exemplo abaixo demonstram os resultados das funções dplyr::left_join() e dplyr::right_join() para um versão reduzida dos dados clima_rs_tbl.
clima_rs_tbl_mini <- clima_rs_tbl %>%
slice(., 1:3)
clima_rs_tbl_mini
# combina os dados baseado nas observações dos dados à esquerda (x)
left_join(
x = clima_rs_tbl_mini,
y =metadados_rs,
by = "codigo"
)
# combina os dados baseado nas observações dos dados à direita (y)
right_join(
x = clima_rs_tbl_mini,
y = metadados_rs,
by = "codigo"
)